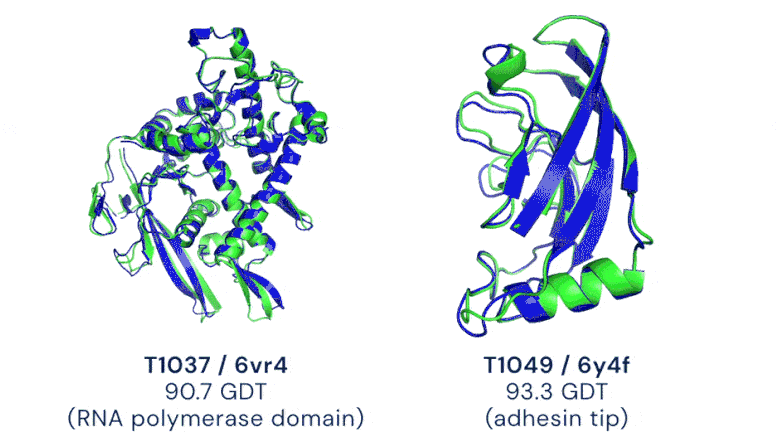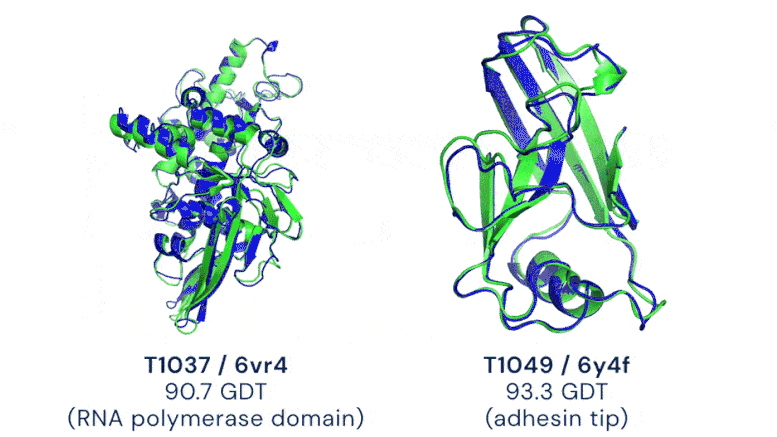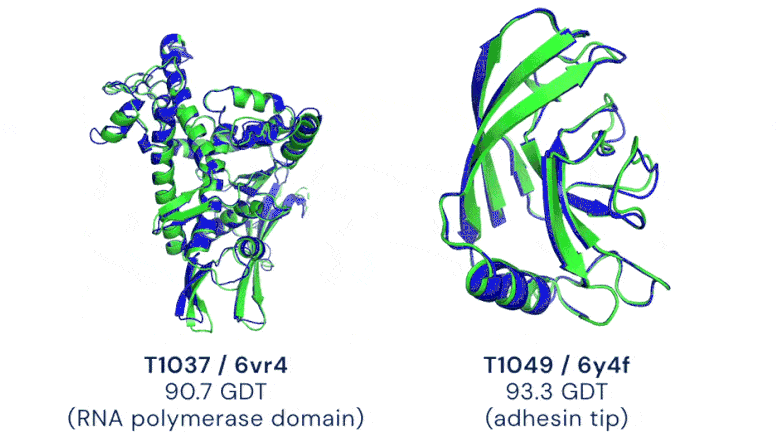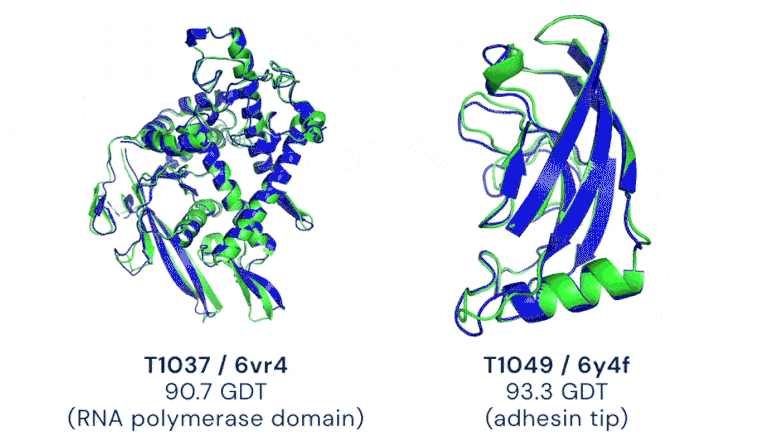
Zwei Beispiele für Proteinziele in der Kategorie der freien Modellierung. AlphaFold sagt hochgenaue Strukturen voraus, die anhand des experimentellen Ergebnisses gemessen werden. Bildnachweis: DeepMind
DeepMind entwickelt eine KI-Lösung für die 50 Jahre alte Proteinherausforderung und schafft so das Potenzial, die biologische Forschung zu beschleunigen.
In einem großen wissenschaftlichen Fortschritt wurde die neueste Version des KI-Systems AlphaFold von DeepMind als Lösung für die 50 Jahre alte große Herausforderung der Vorhersage der Proteinstruktur anerkannt, die laut einer strengen unabhängigen Studie häufig als “Proteinfaltungsproblem” bezeichnet wird Bewertung. Dieser Durchbruch könnte die biologische Forschung langfristig erheblich beschleunigen und unter anderem neue Möglichkeiten für das Verständnis von Krankheiten und die Entdeckung von Arzneimitteln eröffnen.
Die Ergebnisse von CASP14 zeigen heute, dass das neueste AlphaFold-System von DeepMind ein beispielloses Niveau erreicht Richtigkeit in der Strukturvorhersage. Das System ist in der Lage, innerhalb weniger Tage hochgenaue Strukturen zu bestimmen. CASP, die kritische Bewertung der Vorhersage der Proteinstruktur, ist eine alle zwei Jahre von der Community durchgeführte Bewertung, die 1994 gestartet wurde und der Goldstandard für die Bewertung von Vorhersagetechniken ist. Die Teilnehmer müssen blind die Struktur von Proteinen vorhersagen, die erst kürzlich – oder in einigen Fällen noch nicht – experimentell bestimmt wurden, und warten, bis ihre Vorhersagen mit experimentellen Daten verglichen werden.
CASP verwendet die Metrik „Global Distance Test (GDT)“, um die Genauigkeit zwischen 0 und 100 zu bewerten. Das neue AlphaFold-System erreicht über alle Ziele hinweg einen Medianwert von 92,4 GDT. Der durchschnittliche Fehler des Systems beträgt ungefähr 1,6 Angström – ungefähr die Breite eines Atom. Laut Professor John Moult, Mitbegründer und Vorsitzender von CASP, wird eine Punktzahl von rund 90 GDT informell als wettbewerbsfähig mit Ergebnissen aus experimentellen Methoden angesehen.
Professor John Moult, Mitbegründer und Vorsitzender von CASP, University of Maryland, sagte:
„Wir beschäftigen uns seit fast 50 Jahren mit diesem einen Problem – wie falten sich Proteine zusammen? Es ist ein ganz besonderer Moment, zu sehen, wie DeepMind eine Lösung für dieses Problem erarbeitet, nachdem wir so lange persönlich an diesem Problem gearbeitet haben und uns nach so vielen Zwischenstopps gefragt haben, ob wir jemals dort ankommen würden. “
Warum die Vorhersage der Proteinstruktur wichtig ist
Proteine sind lebenswichtig und ihre Formen sind eng mit ihren Funktionen verbunden. Die Fähigkeit, Proteinstrukturen genau vorherzusagen, ermöglicht ein besseres Verständnis dessen, was sie tun und wie sie funktionieren. Derzeit befinden sich über 200 Millionen Proteine in der Hauptdatenbank, und nur ein Bruchteil ihrer 3D-Strukturen wurde kartiert.
Eine große Herausforderung ist die astronomische Anzahl von Möglichkeiten, wie sich ein Protein theoretisch falten könnte, bevor es sich in seine endgültige 3D-Struktur einfügt. Viele der größten gesellschaftlichen Herausforderungen, wie die Entwicklung von Therapien für Krankheiten oder die Suche nach Enzymen, die Industrieabfälle abbauen, hängen im Wesentlichen mit Proteinen und ihrer Rolle zusammen. Die Bestimmung von Proteinformen und -funktionen ist ein wichtiges Gebiet der wissenschaftlichen Forschung, in erster Linie unter Verwendung experimenteller Techniken, die jahrelange mühsame und mühsame Arbeit pro Struktur erfordern können und die Verwendung von Spezialgeräten im Wert von mehreren Millionen Dollar erfordern.
DeepMinds Ansatz zum Problem der Proteinfaltung
Dieser Durchbruch baut auf DeepMinds erstem Eintrag bei CASP13 im Jahr 2018 auf, bei dem die erste Version von AlphaFold unter allen Teilnehmern die höchste Genauigkeit erreichte. Jetzt hat DeepMind neue Deep-Learning-Architekturen für CASP14 entwickelt, die sich von den Bereichen Biologie, Physik und maschinelles Lernen sowie von der Arbeit vieler Wissenschaftler auf dem Gebiet der Proteinfaltung im letzten halben Jahrhundert inspirieren lassen.
Ein gefaltetes Protein kann als „räumlicher Graph“ betrachtet werden, bei dem Reste die Knoten sind und Kanten die Reste in unmittelbarer Nähe verbinden. Dieser Graph ist wichtig für das Verständnis der physikalischen Wechselwirkungen innerhalb von Proteinen sowie ihrer Evolutionsgeschichte. Für die neueste Version von AlphaFold, die bei CASP14 verwendet wird, hat DeepMind ein auf Aufmerksamkeit basierendes neuronales Netzwerksystem erstellt, das durchgängig trainiert wird und versucht, die Struktur dieses Diagramms zu interpretieren, während es über das implizite Diagramm nachdenkt, das es erstellt. Es verwendet evolutionär verwandte Sequenzen, Multiple Sequence Alignment (MSA) und eine Darstellung von Amino Acid Restpaare zur Verfeinerung dieses Diagramms.
Durch Iteration dieses Prozesses entwickelt das System starke Vorhersagen über die zugrunde liegende physikalische Struktur des Proteins. Zusätzlich kann AlphaFold mithilfe eines internen Konfidenzmaßes vorhersagen, welche Teile jeder vorhergesagten Proteinstruktur zuverlässig sind.
Das System wurde auf öffentlich verfügbaren Daten trainiert, die aus ~ 170.000 Proteinstrukturen aus der Proteindatenbank bestehen, wobei für moderne Standards des maschinellen Lernens ein relativ bescheidener Rechenaufwand verwendet wurde – ungefähr 128 TPUv3-Kerne (ungefähr gleich 100-200 GPUs) werden überfahren ein paar Wochen.
Potenzial für reale Auswirkungen
DeepMind freut sich darauf, mit anderen zusammenzuarbeiten, um mehr über das Potenzial von AlphaFold zu erfahren, und das AlphaFold-Team untersucht, wie Vorhersagen der Proteinstruktur mit einigen Fachgruppen zum Verständnis bestimmter Krankheiten beitragen können.
Es gibt auch Anzeichen dafür, dass die Vorhersage der Proteinstruktur bei zukünftigen Bemühungen zur Reaktion auf Pandemien als eines von vielen von der wissenschaftlichen Gemeinschaft entwickelten Instrumenten nützlich sein könnte. Anfang dieses Jahres sagte DeepMind mehrere Proteinstrukturen der SARS-CoV-2 Das Virus und die beeindruckend schnelle Arbeit von Experimentatoren haben nun bestätigt, dass AlphaFold ein hohes Maß an Genauigkeit bei seinen Vorhersagen erreicht hat.
AlphaFold ist einer der bedeutendsten Fortschritte von DeepMind. Aber wie bei allen wissenschaftlichen Forschungen gibt es noch viel zu tun, einschließlich herauszufinden, wie mehrere Proteine Komplexe bilden und wie sie miteinander interagieren DNA, RNAoder kleine Moleküle und wie man die genaue Position aller Aminosäureseitenketten bestimmt.
Wie bei seinem früheren CASP13 AlphaFold-System plant DeepMind, zu gegebener Zeit ein Papier über die Funktionsweise dieses Systems in einem von Experten begutachteten Journal einzureichen und gleichzeitig zu untersuchen, wie ein breiterer Zugriff auf das System auf skalierbare Weise am besten ermöglicht werden kann.
AlphaFold geht neue Wege, um das erstaunliche Potenzial der KI als Hilfsmittel für grundlegende wissenschaftliche Entdeckungen zu demonstrieren. DeepMind freut sich auf die Zusammenarbeit mit anderen, um dieses Potenzial auszuschöpfen.
Aussagen unabhängiger Wissenschaftler:
Professor Venki Ramakrishnan, Nobelpreisträger und Präsident der Royal Society
„Diese Computerarbeit stellt einen erstaunlichen Fortschritt in Bezug auf das Problem der Proteinfaltung dar, eine 50 Jahre alte große Herausforderung in der Biologie. Es ist Jahrzehnte her, bevor viele Leute auf dem Gebiet vorausgesagt hätten. Es wird spannend zu sehen sein, auf welche Weise die biologische Forschung grundlegend verändert wird. “
Professor Dame Janet Thornton, emeritierte Direktorin und leitende Wissenschaftlerin, EMBL-EBI
„Was das DeepMind-Team erreicht hat, ist fantastisch und wird die Zukunft der Strukturbiologie und Proteinforschung verändern. Nachdem ich jahrzehntelang Proteine untersucht hatte, die Moleküle, die die Struktur und Funktionen aller Lebewesen bereitstellen, erwachte ich heute Morgen mit dem Gefühl, dass Fortschritte erzielt wurden. “
Arthur D. Levinson, PhD, Gründer und CEO Calico, ehemaliger Vorsitzender und CEO von Genentech
„AlphaFold ist ein einmaliger Fortschritt, der Proteinstrukturen mit unglaublicher Geschwindigkeit und Präzision vorhersagt. Dieser Sprung nach vorne zeigt, wie Computermethoden die Forschung in der Biologie verändern und vielversprechend sind, um den Prozess der Wirkstoffentdeckung zu beschleunigen. “
Professor Andrei Lupas, Direktor des Max-Planck-Instituts für Entwicklungsbiologie
“Mit den erstaunlich genauen Modellen von AlphaFold konnten wir eine Proteinstruktur lösen, an der wir fast ein Jahrzehnt lang festgehalten hatten, und unsere Bemühungen, zu verstehen, wie Signale über Zellmembranen übertragen werden, neu starten.”
Professor Ewan Birney, stellvertretender Generaldirektor EMBL, Direktor EMBL-EBI
„Ich wäre fast von meinem Stuhl gefallen, als ich diese Ergebnisse sah. Ich weiß, wie streng CASP ist – es stellt im Grunde sicher, dass Computermodelle die herausfordernde Aufgabe der Ab-initio-Proteinfaltung erfüllen müssen. Es war demütig zu sehen, dass diese Modelle das so genau können. Es wird viele Aspekte zu verstehen geben, aber dies ist ein großer Fortschritt für die Wissenschaft. “
Aussagen von DeepMind / Alphabet:
Demis Hassabis, PhD, Gründer und CEO von DeepMind
„Die ultimative Vision hinter DeepMind war es immer, KI aufzubauen und sie dann zu nutzen, um unser Wissen über die Welt um uns herum zu erweitern, indem das Tempo der wissenschaftlichen Entdeckung beschleunigt wird. Für uns ist AlphaFold ein erster Beweis für diese These. Dieser Fortschritt ist unser erster großer Durchbruch in einer langjährigen großen Herausforderung in der Wissenschaft, von der wir hoffen, dass sie einen großen Einfluss auf das Krankheitsverständnis und die Wirkstoffentdeckung in der Praxis haben wird. “
Pushmeet Kohli, PhD, Leiter AI for Science, DeepMind
„Diese unglaublichen Ergebnisse sind ein Beweis für die einzigartige Forschungsphilosophie von DeepMind: Missionsorientierte, multidisziplinäre Teams zusammenzubringen, um ehrgeizige wissenschaftliche Ziele zu erreichen. Kritische Bewertungen wie CASP sind wichtig, um den Forschungsfortschritt voranzutreiben, und wir freuen uns darauf, auf dieser Arbeit aufzubauen, unser Verständnis von Proteinen und biologischen Mechanismen zu vertiefen und neue Wege der Erforschung zu eröffnen. “
John Jumper, PhD, AlphaFold Lead, DeepMind
„Die Proteinbiologie ist fantastisch komplex und lässt sich nicht einfach charakterisieren. Die Arbeit unseres Teams zeigt, dass Techniken des maschinellen Lernens endlich in der Lage sind, die Komplexität der Beschreibung dieser unglaublichen Proteinmaschinen zu bewältigen, und wir sind wirklich gespannt, welche neuen Durchbrüche sowohl in der menschlichen Gesundheit als auch in der grundlegenden Biologie daraus resultieren werden. “
Kathryn Tunyasuvunakool, PhD, Wissenschaftsingenieurin, DeepMind
„Die Fähigkeit, hochgenaue Proteinstrukturen mit KI vorherzusagen, könnte unsere Herangehensweise an die Biologie verändern und potenzielle Anwendungen für das Wirkstoffdesign und die Bioremediation bieten. Insbesondere für experimentell herausfordernde Proteine könnten gute Vorhersagetechniken einen großen Unterschied machen. “
Sundar Pichai, CEO, Google und Alphabet
„Dies ist ein unglaublicher KI-gestützter Durchbruch bei der Proteinfaltung, der uns helfen wird, einen der grundlegendsten Bausteine des Lebens besser zu verstehen. Dieser enorme Fortschritt von DeepMind hat unmittelbare praktische Auswirkungen und ermöglicht es Forschern, neue und schwierige Probleme anzugehen, von der zukünftigen Reaktion auf eine Pandemie bis hin zur ökologischen Nachhaltigkeit. “
Lesen Sie die DeepMind AI-Lösung für eine 50 Jahre alte wissenschaftliche Herausforderung, die die medizinische Forschung revolutionieren könnte, um mehr zu diesem Thema zu erfahren.
Über DeepMind
DeepMind ist ein multidisziplinäres Team von Wissenschaftlern, Ingenieuren, Experten für maschinelles Lernen und mehr, das zusammenarbeitet, um sichere KI-Systeme zu erforschen und aufzubauen, die lernen, Probleme zu lösen und wissenschaftliche Entdeckungen für alle voranzutreiben.
DeepMind ist bekannt für die Entwicklung von AlphaGo, dem ersten Programm, das einen Weltmeister im komplexen Go-Spiel besiegt hat. Es hat über 1000 Forschungsarbeiten veröffentlicht – darunter mehr als ein Dutzend in Natur und Wissenschaft – und in vielen herausfordernden KI-Bereichen bahnbrechende Ergebnisse erzielt StarCraft II zur Proteinfaltung.
DeepMind wurde 2010 in London gegründet und hat sich 2014 mit Google zusammengetan, um seine Arbeit zu beschleunigen. Seitdem hat sich die Community um Teams in Alberta, Montreal, Paris und Mountain View in Kalifornien erweitert.